MUM
From CSBLwiki
연구 목표
- 유전자 수준에서 여러 유전체 서열을 동시 비교 분석하여 유전자 클러스터를 발굴하는 알고리즘 개발하여 새로운 클러스터 예측방법 제시
- 두가지 종류의 균류 유전체 데이터베이스에 적용하여 클러스터 데이터 베이스 구축 및 여러 유전체 서열에서 보존되어 나타나는 유전자 클러스터에 대한 탐구(1.클러스터 데이터 베이스를 통한 생물 간의 계통적 관계 제시 2. 클러스터 내 유전자 사이의 연관성 연구 3. 특정 기능에 관여하는 유전자 클러스터 발굴 등)
실험에 사용된 데이터 베이스
1. NCBI에서 제공하는 16종의 균류 유전체 서열
2. JGI에서 시퀀싱 되고 어노테이션 된 206종의 균류 유전체 서열
연구 계획
실험 방법
KO mum algorithm
기존의 clustering algorithm 들은 multiple sequence alignment (MSA) 에 기반하여 cluster를 찾아낸다. 그러나 단순히 서열간의 분석으로서 유전자의 기능을 고려 하지 않는다. 이러한 단점을 극복하기 위하여 본연구는 유전자의 기능에 기반하여 유전체를 비교해서 cluster 를 찾아내는 ko mum algorithm 을 개발 하였다. 다른 생물로부터 유래한 유전자를 비교하기 위해 KEGG Orthology (KO)/clusters of euKaryotic Orthologous Groups (KOG) 등의 데이터베이스를 사용하여 유전자를 annotation 하고, 그 순서를 비교하여 동일한 순서로 발견되는 유전자를 찾고 이를 KO mum로 이름지었다. 본 연구에서는 KO mum 알고리즘의 개발과 함께 예측된 ko mum을 분석하여 생물학적 의미를 탐색하였다.
KO mum 알고리즘을 개발하고 실험하기 위하여 실험데이터로 NCBI에서 제공하는 16종의 균류 유전체 서열을 이용하였으며 3027개의 mum을 획득하였고 획득된 데이터를 분석하기 위한 추가 실험을 시행 하였으며 두번째 실험에서는 JGI에서 제공하는 206개의 유전체 서열을 분석 하였다.
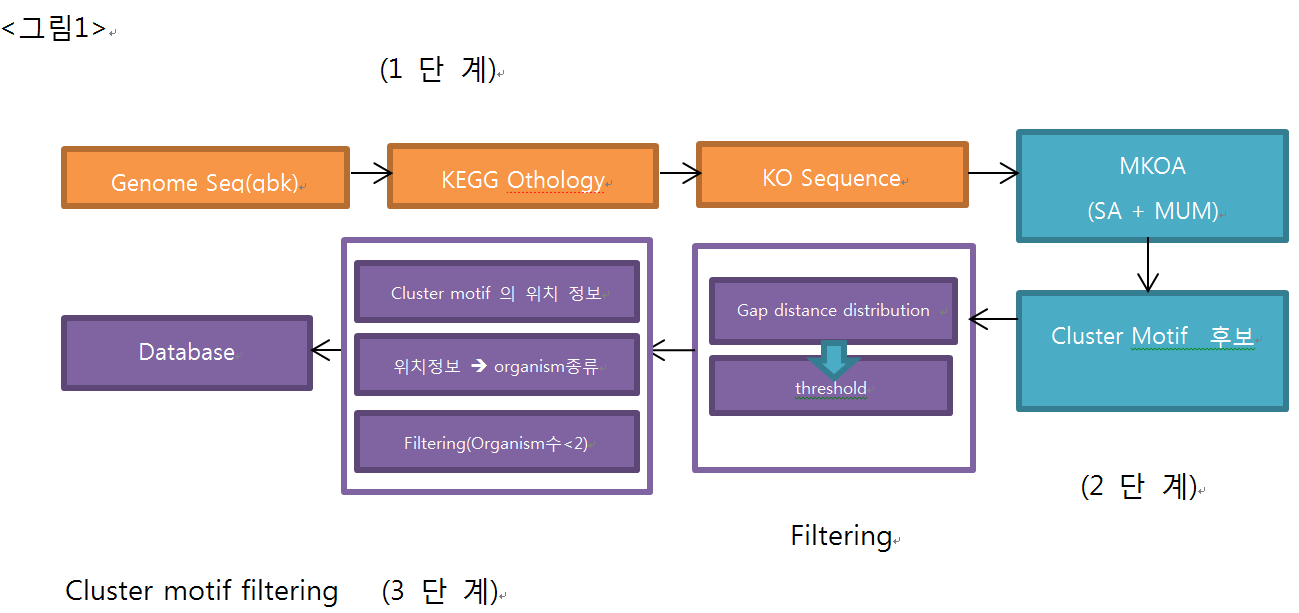
KO MUM algorithm 의 구성은 다음 그림과 같으며 크게 3단계로 나뉘어진다.
1단계
1단계에서는 먼저 유전체의 유전자들을 Orthology database로 annotation한 후 유전체 서열을 어노테이션된 orthology id 로 변환된 문자열 서열로 변환한다. 또한 어노테이션되지 못한 유전자는 gap으로 정의하며 해당 유전자의 위치정보 및 유전자 길이를 확인하기 위하여 NOnorthologous gene을 줄여 "NO"로 표기하며 NO의 앞과 뒤에 있는 KO/KOG의 위치정보를 이용하여 gap의 길이를 확인하고 전체 유전자의 평균적인 길이인 2000으로 나눈 후 반올림 하여 1부터 9까지의 정수로 변환시켜 문자열 "NO"의 뒤에 추가한 후 문자열 서열에 넣는다. 또한 1단계에서는 각 유전자의 단백질 도메인정보와 orthologous gene database에서 제공하는 기능 클래스 정보, 유전체상에서의 위치정보를 서로 연결하여 다음 2, 3단계에서 불러올 수 있도록 처리한다.
2단계
변환된 문자열 서열을 비교 분석하여 공통적인 문자열인 mum을 찾기 위하여 suffix array algorithm을 사용하여 전체 서열의 자료구조를 만들었으며 구성된 자료구조를 재분석하여 mum후보를 추출하기 위한 알고리즘을 추가적으로 실행하였다. 추가적으로 만들어진 알고리즘은 2가지 조건을 이용하여 suffix array로 부터 얻은 공통적인 문자열을 재분석한다.
예를 들어 다음 4가지 문자열이 있다고 했을때,
S1 = A,B
S2 = A,B,C,D
S3 = A,B,C,F
S4 = A,B,C,D,F
우리가 실제로 찾아야할 공통된 문자열은 다음과 같다.
A,B = S1,S2,S3,S4
A,B,C = S2,S3,S4
ABCD = S2,S4
그런데 suffix array를 통해 나온 문자열은 다음과 같으며 중복되어 출력된다.
A = S1,S2,S3,S4
AB = S1,S2,S3,S4
ABC = S1,S2,S3,S4
ABCD = S2,S4
따라서 첫번째로 공통된 문자열 중 중복되어 나타나는 하위 문자열을 삭제해야한다( 예 A는 AB에 포함되어 삭제, AB는 ABC에 포함되어 삭제...). 그러나 여기서 AB는 ABC에 속해 있지만 AB(S2,S3,S4)는 삭제해선 안된다.S1에 의해 또 하나의 mum이 되기 때문이다. 2단계에서는 이렇게 이 두가지 조건을 고려하여 mum 후보를 예측한다.
3단계
3단계는 mum후보를 필터링하는 과정으로 2개의 조건에 따라 필터링한다.
먼저 gap으로 정의된 NO의 유전자길이에 따라 mum이 될 수 있는가 없는가를 판단하는 과정으로 임의적인 설정을 통해 gap의 길이가 너무 길때는 하나의 mum으로 판단하지 않고 대신 NO를 사이에 둔 양쪽 KO/KOG 문자열을 mum으로 고려할 수 있는지를 판단한 후 mum을 쪼갠다. 또한 mum의 맨 앞, 맨뒤에 NO가 존재할 경우 NO를 삭제한 후의 mum에 속한 유전자의 수를 판단하여 너무 작을 경우(1개~2개?) mum으로 판단하지 않았다.
두번째로 Fungi의 genome을 살펴보니 같은 gene이 genome상에서 여러번 반복해서 존재하는 경우가 굉장히 많았고 때문에 하나의 유전체에서 공통된 mum 후보가 생기는 경우가 있었다. 그래서 mum의 유전체상에서 위치정보를 통해 하나의 유전체 상에서만 존재하는 mum후보를 분별하여 삭제하였다.
JGI genome 연구 결과
KOG cluster
1.JGI에서 206종의 유전체 서열을 다운로드한 후 KOG로 어노테이션 된 문자열 서열로 변환함 2.전체 유전체 서열의 50퍼센트 이상은 서열의 50% -78% 이상 KOG로 어노테이션 되었음 자료 >> File:JGI genes.xls
3.예측된 총 클러스터의 개수는 94240개임
각 유전체의 클러스터 자료 >> File:JGI cluster database.txt
4. 유전자 클러스터의 프로화일 완료
<추가 > 5. KOG로 구성된 클러스터의 interpro 변환 및 기능 클래스 확인
Interpro
1. JGI에서 Interpro 어노테이션 정보 다운로드
2. 유전체 정보의 커버 정도 확인 및 데이터정보 확인 완료
3. Interpro를 어떻게 사용할 것 인가에 대한 공부중