MUM
From CSBLwiki
(→KO mum algorithm) |
|||
| Line 27: | Line 27: | ||
== 실험 방법 == | == 실험 방법 == | ||
=== KO mum algorithm === | === KO mum algorithm === | ||
| - | [[File:MUM method.png| | + | [[File:MUM method.png|800px|그림1]] |
| Line 49: | Line 49: | ||
: Fungi의 genome을 살펴보니 같은 gene이 genome상에서 여러번 반복해서 존재하는 경우가 굉장히 많았고 때문에 한 genome상에서 cluster를 가지는 경우가 있었다. 그래서 cluster의 genome상에서 위치 정보를 추출한 후 organism정보를 얻어 이 정보를 통해 분별해 냈다. | : Fungi의 genome을 살펴보니 같은 gene이 genome상에서 여러번 반복해서 존재하는 경우가 굉장히 많았고 때문에 한 genome상에서 cluster를 가지는 경우가 있었다. 그래서 cluster의 genome상에서 위치 정보를 추출한 후 organism정보를 얻어 이 정보를 통해 분별해 냈다. | ||
| - | |||
== JGI genome 연구 결과 == | == JGI genome 연구 결과 == | ||
Revision as of 07:21, 20 May 2013
< MUM algorithm 및 포스터 계획>
|
연구 목표
Clustering algorithm 은 서로 다른 genome sequence 서열을 비교 분석 할 수 있는 multiple alignment를 사용 하여 Cluster를 찾아낸다. 그러나 기존의 algorithm들은 단순히 sequence 서열간의 분석으로서 gene의 기능을 고려 하지 않는다는 단점이 있다. 그래서 sequence 간의 비교 분석과 함께 gene 기능정보를 제공 하는 KEGG database를 이용하여 genome을 비교 분석 하여 cluster 를 찾아내는 KO MUM algorithm을 개발 하였다. Raw data로 whole genome을 가지는 총 16종 fungi를 선택 하였다.
연구 계획
-KO MUM algorithm 개발과 함께 algorithm의 정확성과 타당성을 확인하기 위하여 여러 접근 방식으로 Fungi cluster를 분석한다.
- 0. cluster database 구축
- 1. 보존된 Cluster는 일반적으로 유사한 기능을 하거나 특정 역할을 함께 하는 gene으로 구성된다고 알려져있다. 따라서 각 cluster 내의 KO를 KEGG 에서 제공하는 6가지 대분류로 분류 하고 cluster의 class를 계산 하는 것 으로 MUM cluster내 gene 사이의 기능적 유사성을 평가 할 수 있으며 유사성에 대한 분석을 통해 algorithm의 타당성을 판단 하는데 도움이 될 수 있을 것 이다.
- 2. cluster는 KEGG orthology 로 annotation 된 gene 뿐 아니라 annotation 되지 못하는 gene(NA gene)을 포함 하고 있으며, cluster 가 포함 되는 각 genome 상의 NA gene을 확인 해본 결과
몇 몇 NA gene 은 orthology는 아니더라도 같은 fpam domain을 포함 하고 있다는 것 을 확인 하였다. 얼마나 많은 cluster의 NA gene이 같은 domain을 가지고 있는지 계산을 할 것 이다. 계산을 통해서 1번의 자료를 뒷받침 할 수 있을 것 이다.
- 3. 보존된 cluster를 가지고 있다는 것은 진화적으로 근접하다는 것으로 생각된다.각 fungi의 cluster를 profiling 하고 profile을 통해서 계통도를 계산 할 것 이다.
- 4. Network ( Network는 포스터에서 제외 할 생각)
실험 방법
KO mum algorithm
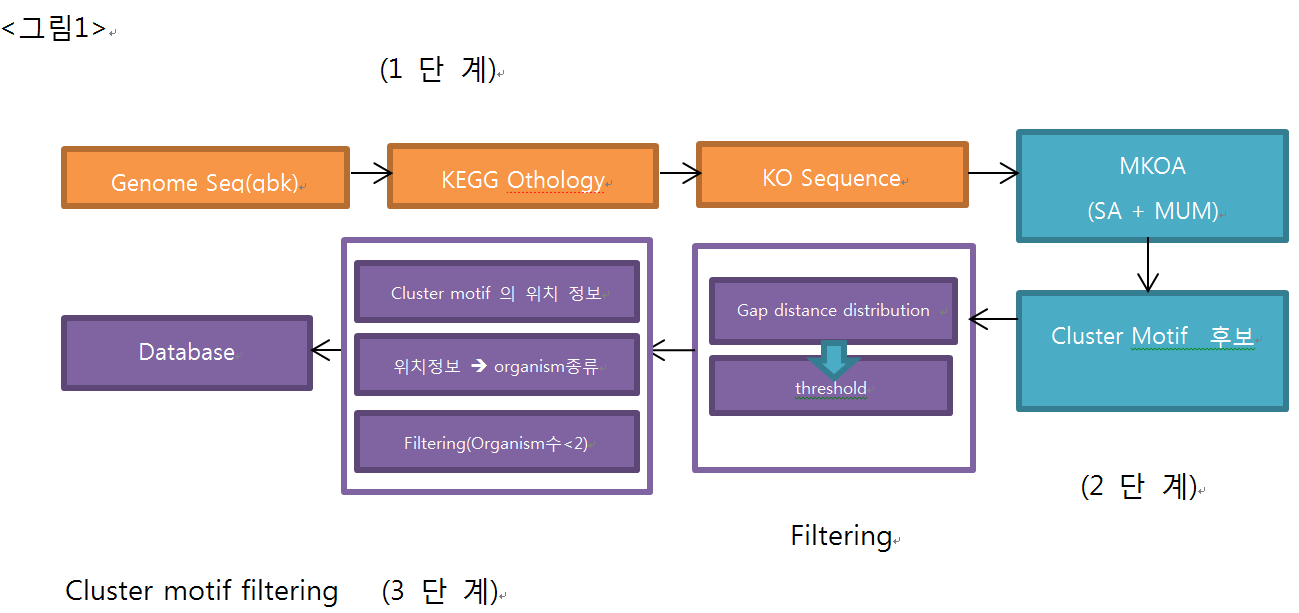
- 1단계 : genome sequence > KO string으로 변환 하는 과정
- genome sequence를 KEGG Orthology(KO)database 를 사용하여 annotation한다. KO는 문자열로써 genome sequence는 KO string으로 변환되어진다.
- 2단계 : KO string > KO mum algorithm > MUM cluster
- KO string을 비교 분석 하여 cluster 후보를 선별 하는 과정으로 일반적으로 multiple sequence alignment algorithm 중 하나인 Suffix array 를 사용하는 KO mum algorithm을 이용하여 MUM cluster 후보를 추출한다.
- 3-1 단계: genome seq > gap & organism에서 cluster의 위치 정보 추출 > 유효한 cluster 판별 과정
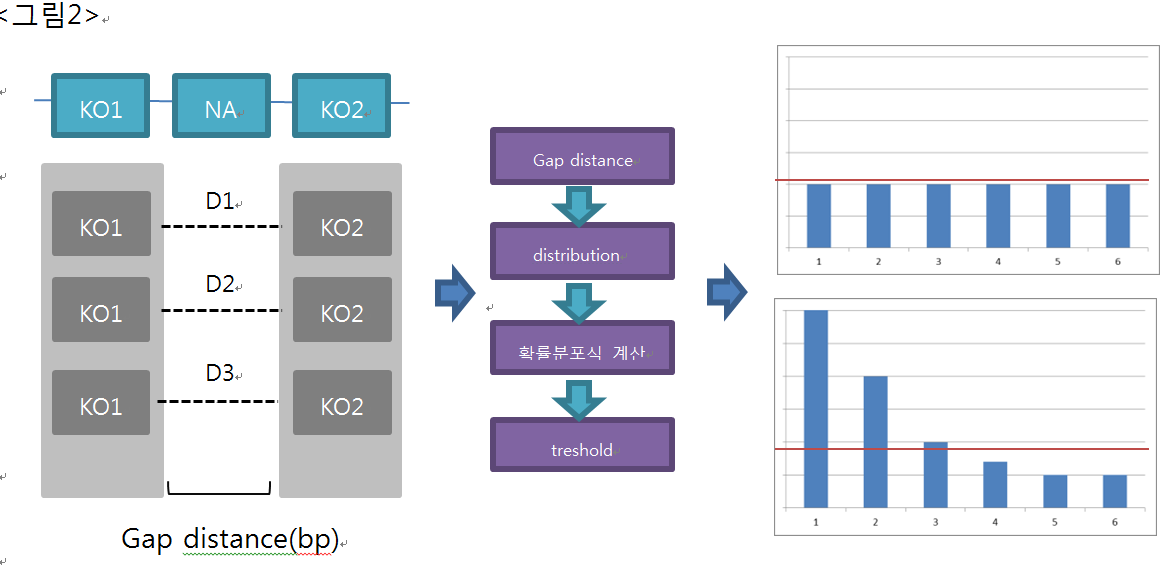
- Cluster가 존재하는 organism을 살펴보면 NA gene이 존재하며 NA gene이 존재 하는 경우 NA gene 양쪽의 KO 사이의 거리(gap) 는 크게 차이 날 수있다. 이러한 경우 cluster 내의 KO 순서가 정확히 같다고 판단하기 어렵기 때문에 KO와 KO 사이의 gap(KO의 끝지점 부터 다음 KO의 시작 지점)을 고려하여 선출된 cluster 후보가 유효한지 판별하는 과정이 필요하다.
- 이 과정은 genome sequence로부터 모든 gap 의 정보를 얻고 각 organism 에서 cluster의 위치정보를 추출한다. 그 후 각각 위치에서 Cluster 내 KO의 gap distance 의 확률분포를 계산 하여 threshold(99%)를 구한다(그림2). 그 후 Threshold에 따라 Cluster를 평가 하여 유효한 Cluster 인지 확인 한다.
- 3-2 단계: 한 organism에서만 존재하는 cluster 제거 과정
- Fungi의 genome을 살펴보니 같은 gene이 genome상에서 여러번 반복해서 존재하는 경우가 굉장히 많았고 때문에 한 genome상에서 cluster를 가지는 경우가 있었다. 그래서 cluster의 genome상에서 위치 정보를 추출한 후 organism정보를 얻어 이 정보를 통해 분별해 냈다.
JGI genome 연구 결과
NCBI genome 연구 결과
cluster database 구축
16종의 fungi 에서 총 3076개의 클러스터를 얻었으며 각 fungi 의 cluster 중 여러 종에 함께 포함된 cluster의 개수는 다음과 같다.
Zygosaccharomyces_rouxii_CBS_732_uid39573 1018
Pichia_stipitis_CBS_6054_uid18881 589
Debaryomyces_hansenii_CBS767_uid12410 604
Candida_dubliniensis_CD36_uid38659 547
Eremothecium_gossypii_uid10623 1055
Lachancea_thermotolerans_CBS_6340_uid39575 1057
Candida_glabrata_CBS138_uid12376 606
Kluyveromyces_lactis_NRRL_Y-1140_uid12377 910
Saccharomyces_cerevisiae_uid128 743
Pichia_pastoris_GS115_uid39439 355
Aspergillus_fumigatus_uid14003 23
Yarrowia_lipolytica_CLIB122_uid12414 149
Candida_albicans_uid14005 23
Schizosaccharomyces_pombe_uid127 15
Cryptococcus_neoformans_var_JEC21_uid10698 3
Encephalitozoon_cuniculi_uid155 3
cluster classification
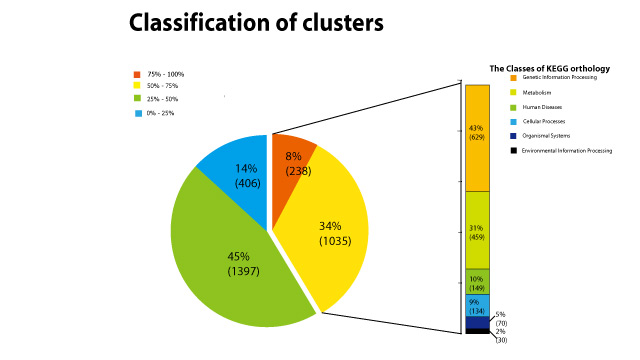
각 클러스터 내의 KO 의 Class 의 일치정도를 확인하여 클러스터 내 유전자의 기능이 서로 어느 정도 유사한지 확인한다. 전체 유전자 중 일치하는 유전자 비율에 따라 cluster를 나누었다. 대부분의 cluster는 3~ 4개의 유전자를 포함 하고 있으며 이 유전자들 중 1개 이상은 NO 인경우가 많으므로 75~ 100% 를 완전하게 일치하는 cluster로 분류 하였고, 50% 이상의 일치도를 보이는 cluster 만을 최소 단위로 인정 하여 이 클러스터의 클래스 분포를 확인 하였다. 클러스터의 클래스 분포를 살펴보니 대부분 cluster는 genetic information processing 과 metabolism이었으며 세부적인 기능을 살펴 본다면 기능적인 관련성을 더 잘 살펴볼 수 있을 것 이다.